研究
研究概要
ゲノムはある生物の持つ1セットかつ全ての遺伝情報である。OISTマリンゲノミックス・ユニット(MGU)ではゲノムの解読を基盤にして、(1) 脊索動物の起源と進化に関する比較ゲノム科学的および比較分子発生生物学的研究、(2)サンゴと褐虫藻(Symbiodinium)の共生関係の成立と崩壊、オニヒトデのサンゴ礁に及ぼす影響など、サンゴ礁の保全に関するゲノム科学的研究、(3)動物の特殊機能に関するゲノム科学的研究、(4)全動物門のゲノム解読を目指す研究、などを進めている。
(1)脊索動物の起源と進化に関する比較ゲノム科学的および比較分子発生生物学的研究:
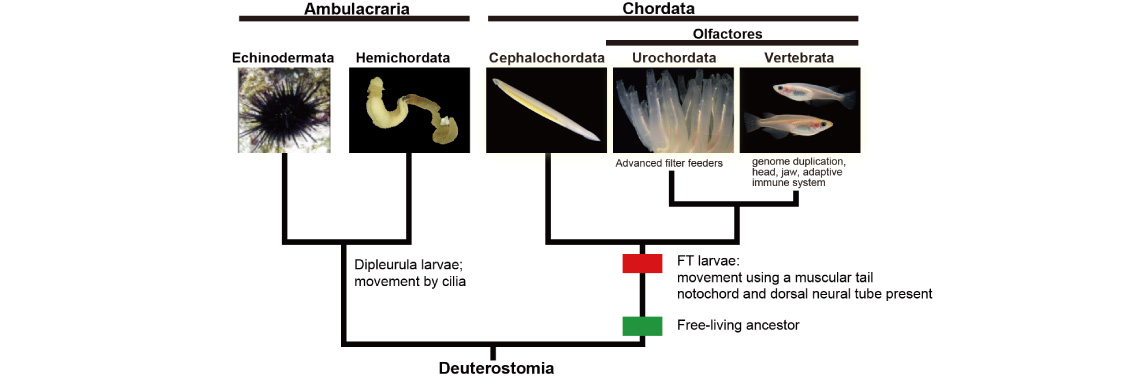
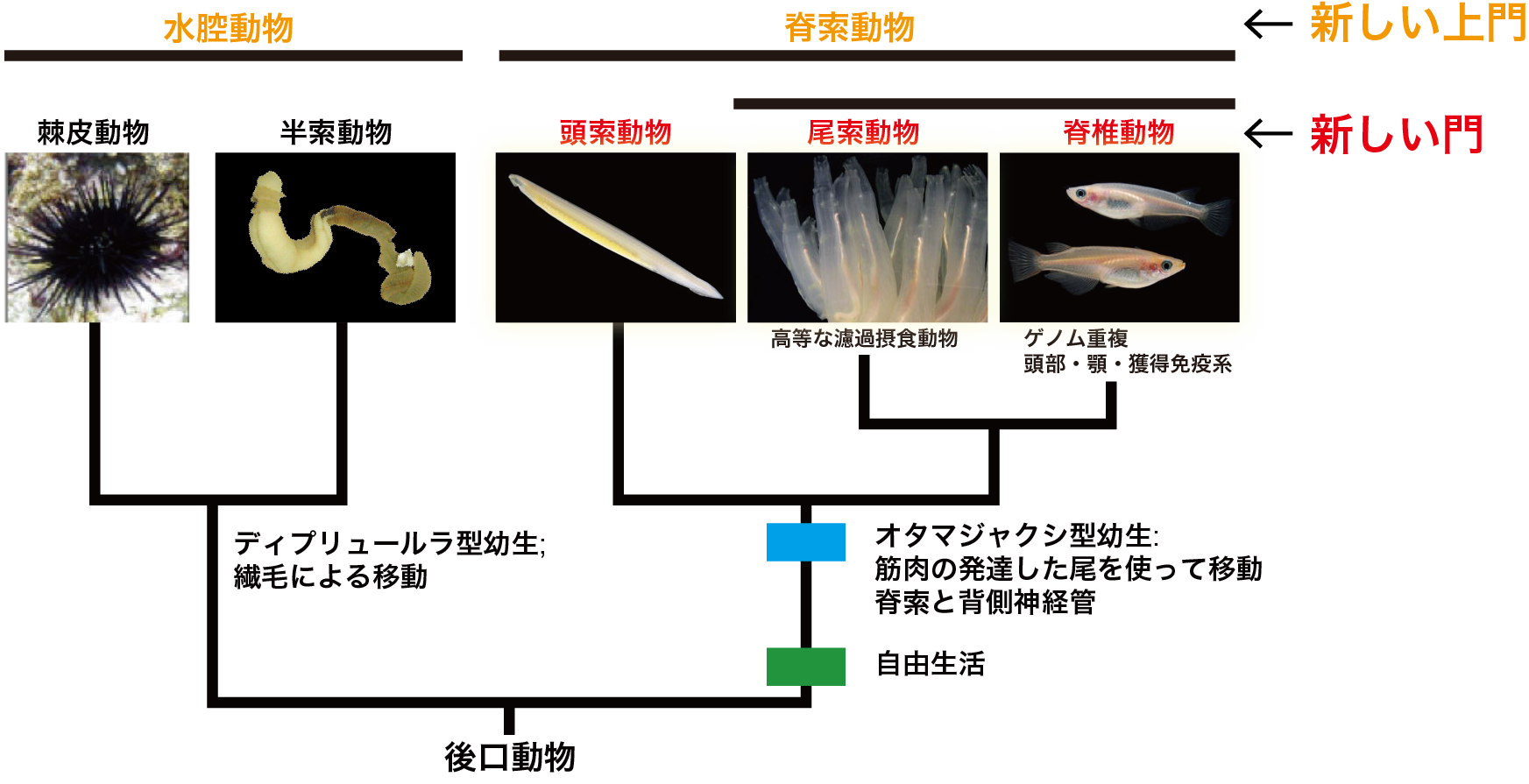
我々脊椎動物はホヤなどの尾索動物とナメクジウオの頭索動物と共に脊索動物と呼ばれ動物群の1員である。これは、この3群が脊索や背側神経管といった明確な共有形質を持つからである。脊索動物はまた、ウニやヒトデなどの棘皮動物、ギボシムシの半索動物と共に新口(後口)動物という大きな動物群を構成する。すなわち、脊索動物は新口動物の共通祖先から、また脊椎動物は脊索動物の共通祖先から進化してきたものと考えられている。我々のユニットでは、脊索動物の起源と進化をゲノム科学的に、また比較分子発生生物学的に解明したいと望んでいる。
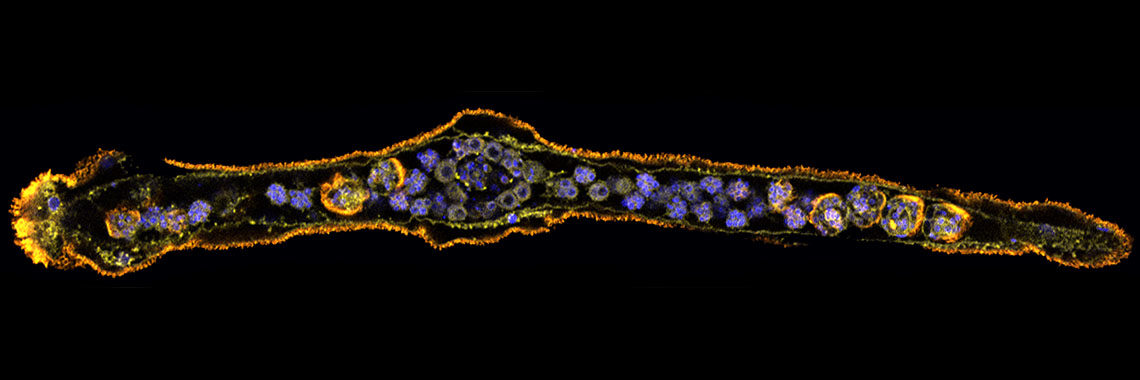
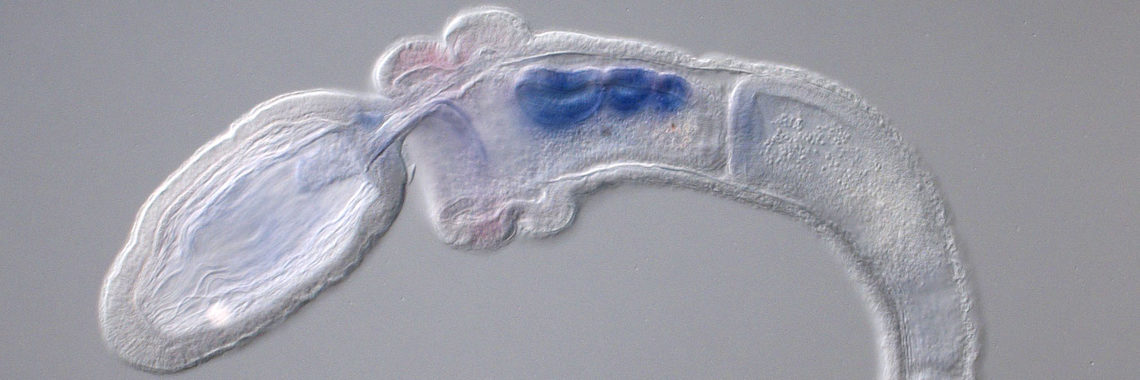
(a) この目的のために、我々はまず2002年にカタユウレイボヤ(Ciona intestinalis)のゲノムを解読した[Dehal et al., Science 298: 2157-2167 (2002) ]。カタユウレイボヤの受精卵は18時間ほどでオタマジャクシ幼生へと発生する。この幼生を構成する細胞の数はわずか2600個ほどであるが、幼生には表皮、背側神経系、消化管、筋肉、脊索、間充織などの細胞が分化し、その体制は脊椎動物を含む脊索動物の体制の基本的でかつ最もシンプルなものと考えられている。解読したカタユウレイボヤのゲノムは約15,600個の遺伝子を含む。我々は、大量のRNA解析と共に、このホヤが持つ、体作りや生命活動の維持で重要な働きをなす転写因子遺伝子とシグナル分子遺伝子にはついて、そのほぼ全ての発現を網羅的に調べ上げた[例えば、Imai et al., Science 312: 1183-1187 (2006): Shoguchi et al., Dev. Biol. 316: 498-509 (2008)]。また、共同研究により、ゲノム情報を駆使したマイクロアレイシステムの作成やトランスジェニック系統の確立とその利用などの基盤整備に貢献した。カタユウレイボヤは現在発生ゲノム科学研究のモデル動物の一つになっている[Satoh, Developmental Genomics of Ascidians, Wiley Blackwell (2014)]。
 ホヤのゲノム解読はまた脊索動物の起源と進化について幾つかの重要な示唆を与えている。一つは尾索動物群が頭索動物と脊椎動物からかなり離れた独自の進化を遂げていること[Satoh (2008)はこれを、尾索動物群のadvanced filter-feeder 仮説として議論している]、その主たる原因がこの動物群を特徴付ける形質である被嚢であり、その成分としてのセルロースを動物で唯一合成できることである。ゲノム解読は、ホヤのセルロース合成酵素遺伝子(CesA)がバクテリアからの水平伝播 (horizontal gene transfer) によって得られ可能性を支持する。つまり、これまで動物の進化とはその関わりが比較的薄いとされてきた遺伝子の水平伝播の重要な役割が明らかになりつつある。また、この発見は(3)の(a)の機能ゲノム科学の展開と結びついている。
ホヤのゲノム解読はまた脊索動物の起源と進化について幾つかの重要な示唆を与えている。一つは尾索動物群が頭索動物と脊椎動物からかなり離れた独自の進化を遂げていること[Satoh (2008)はこれを、尾索動物群のadvanced filter-feeder 仮説として議論している]、その主たる原因がこの動物群を特徴付ける形質である被嚢であり、その成分としてのセルロースを動物で唯一合成できることである。ゲノム解読は、ホヤのセルロース合成酵素遺伝子(CesA)がバクテリアからの水平伝播 (horizontal gene transfer) によって得られ可能性を支持する。つまり、これまで動物の進化とはその関わりが比較的薄いとされてきた遺伝子の水平伝播の重要な役割が明らかになりつつある。また、この発見は(3)の(a)の機能ゲノム科学の展開と結びついている。
(b) 我々は続いて2008年に、ナメクジウオ(Branchiostoma floridae)のゲノムを日米の共同研究で解読した[Putnam et al., Nature 453: 1064-1071 (2008)]。その結果、脊索動物群の中ではナメクジウオが初めに分岐し、次にホヤと脊椎動物が分岐してきたことが分かった。頭索動物ナメクジウオと脊椎動物の間では遺伝子のシンテニーが高度に保存されおり、ナメクジウオのゲノム断片セットを4倍するとヒトの染色体構成に最もうまく対応できることから、脊椎動物の進化の際に2回のゲノムワイドな遺伝子重複が起こったこと、およびその元となった原始的な脊索動物染色体構成を推論することができた。
(c) 棘皮動物ウニのゲノムが2006年に解読されているので、半索動物ギボシムシのゲノムが新口動物の中で唯一未解読のものとなった。この動物のゲノム解読は脊索動物の起源を理解する上で必須であり、長い間日米の共同研究で解読プロジェクトを進めてきた。このプロジェクト推進中に、ギボシムシは、ナメクジウオ・ゲノムに存在するものに相当する基本的なHoxクラスターを持つことを明らかにした[Freeman et al., Curr. Biol. 22: 2053–2058 (2012)]。そして2015年、直接発生種のSaccoglossus kowalevskiiと間接発生種のPtychodera flavaのゲノム解読に成功した[Simakov et al., Nature 527: 459-464 (2015)]。ギボシムシゲノムの解読は、半索動物を含む新口動物の咽頭に関連して、その形成に関わる4つの転写因子遺伝子がクラスターをなして存在するという発見につながった。このクラスターは棘皮動物ヒトデにも存在し[(2)の(e)を参照]、新口動物以外には存在しないので、以前に示唆されていた新口動物群の共有形質としての咽頭を支持するゲノム科学的証拠と言える。
 (d)上に述べたように、我々は今、新口動物5群全てのゲノム情報を得たことになる。これらの比較解析さらにはホヤなどの進化発生学的研究をもとに現在次のような研究を展開している。(1) これまでのように、脊索動物を門として、また頭索動物、尾索動物、脊椎動物をその亜門として取り扱うのではなく、これら3つの動物群それぞれを門として扱い、脊索動物を上門として扱うことを提唱した[Satoh et al., Proc. Royal Soc. B, 281: 20141729 (2014) ]。特に尾索動物と脊椎動物については門として扱うに足る十分は根拠がある。(2)脊索動物の起源と進化についてはこれまで多くの議論がなされてきており、最近では特に背腹軸の反転が脊索動物の起源にとって重要な発生イベントであったとする議論が盛んになされている。脊索は脊索動物を特徴づける最も重要な形質である。我々はこれまでにカタユウレイボヤを用いて脊索形成の分子メカニズムをかなりのレベルまで解明してきたが[Satoh et al., Evol. Dev. 14: 56-75 (2012)]、背腹軸の反転だけで脊索という脊索動物の進化をもたらした新奇な器官の形成を説明することが難しい。私はこれまでに、幼生型の変化、すなわち繊毛を使って遊泳する幼生から尾の筋肉を使って遊泳する幼生への変化が脊索動物の起源をもたらした最も重要な発生学的イベントであることを指摘してきた[Satoh, genesis 46: 614-622 (2008)]。つい最近になって、これらの考えを脊索動物の起源に関する new organizers hypothesis としてまとめたが[Satoh, Chordate Origins and Evolution, Academic Press, 2016]、更に考察する必要がある。
(d)上に述べたように、我々は今、新口動物5群全てのゲノム情報を得たことになる。これらの比較解析さらにはホヤなどの進化発生学的研究をもとに現在次のような研究を展開している。(1) これまでのように、脊索動物を門として、また頭索動物、尾索動物、脊椎動物をその亜門として取り扱うのではなく、これら3つの動物群それぞれを門として扱い、脊索動物を上門として扱うことを提唱した[Satoh et al., Proc. Royal Soc. B, 281: 20141729 (2014) ]。特に尾索動物と脊椎動物については門として扱うに足る十分は根拠がある。(2)脊索動物の起源と進化についてはこれまで多くの議論がなされてきており、最近では特に背腹軸の反転が脊索動物の起源にとって重要な発生イベントであったとする議論が盛んになされている。脊索は脊索動物を特徴づける最も重要な形質である。我々はこれまでにカタユウレイボヤを用いて脊索形成の分子メカニズムをかなりのレベルまで解明してきたが[Satoh et al., Evol. Dev. 14: 56-75 (2012)]、背腹軸の反転だけで脊索という脊索動物の進化をもたらした新奇な器官の形成を説明することが難しい。私はこれまでに、幼生型の変化、すなわち繊毛を使って遊泳する幼生から尾の筋肉を使って遊泳する幼生への変化が脊索動物の起源をもたらした最も重要な発生学的イベントであることを指摘してきた[Satoh, genesis 46: 614-622 (2008)]。つい最近になって、これらの考えを脊索動物の起源に関する new organizers hypothesis としてまとめたが[Satoh, Chordate Origins and Evolution, Academic Press, 2016]、更に考察する必要がある。
現在、新口動物5群のゲノムの比較により、脊索や背側神経管や筋節といった新奇の構造の創出に関与した遺伝子ネットワークの解析を進めており、脊索動物の起源と進化の理解を目指している。
参考文献
- Satoh, N. Chordate Origins and Evolution. Academic Press, New Your (2016)
- Satoh, N. Developmental Genomics of Ascidians. Wiley Brackwell, New York (2014).
- Simakov, O., Kawashima, T., Marlétaz, F., Jenkins, J., Koyanagi, R., Mitros, T., Hisata, K., Bredeson, J., Shoguchi, E., Gyoja, F., Yue, J.X., Chen, Y.C., Robert M. Freeman, R.M., Sasaki, A.,Hikosaka-Katayama, T., Sato, A., Fujie, M., Baughman, K.W., Levine, J., Paul Gonzalez, P., Cameron, C., Fritzenwanker, J.H., Pani, A.M., Goto, H., Kanda, M., Arakaki, N., Yamasaki, S., Qu, J., Cree, A., Ding, Y., Dinh, H.H., Dugan, S., Holder, M., Jhangiani, S.N., Kovar, C.L., Lee, S.L., Lewis, L.R., Morton, D., Nazareth, L.V., Okwuonu, G., Santibanez, J., Chen, R., Richards, S., Muzny, D.M., Gillis, A., Peshkin, L., Wu, M., Humphreys, T., Su, Y.H., Putnam, N.H., Schmutz, J., Fujiyama, A., Yu, J.K., Tagawa, K., Worley, K.C., Gibbs, R.A., Kirschner, M.W., Christopher J. Lowe, C.J., Satoh, N., Rokhsar, D.S., Gerhart, J., Hemichordate genomes and deuterostome origins. Nature 527(7579):459-65. (2015)
- Putnam, N.H., et al., The amphioxus genome and the evolution of the chordate karyotype. Nature 453: 1064-1071(2008).
- Dehal, P., et al., The draft genome of Ciona intestinalis: insights into chordate and vertebrate origins. Science 298: 2157-67 (2002)
- Imai, K.S., Levine, M., Satoh, N., and Satou, Y., Regulatory blueprint for a chordate embryo. Science 312: 1183-1187 (2006).
- Shoguchi, E., Hamaguchi, M., and Satoh, N., Genome-wide network of regulatory genes for construction of a chordate embryo. Dev. Biol. 316: 498-509 (2008).
- Satoh N., An aboral-dorsalization hypothesis for chordate origin. Genesis, 46:614-622 (2008)
- Freeman R, Ikuta T, Wu M, Koyanagi R, Kawashima T, Tagawa K, Humphreys T, Fang GC, Fujiyama A, Saiga H, Lowe C, Worley K, Jenkins J, Schmutz J, Kirschner M, Rokhsar D, Satoh N, Gerhart J. Identical Genomic Organization of Two Hemichordate Hox Clusters. Current Biology 22: 2053–2058 (2012).
- Satoh, N., Rokhsar, D., Nishikawa, T., Chordate evolution and the three-phylum system. Proc. R. Soc. B. 281:1794 (2014)
- Satoh, N., Tagawa, K., and Takahashi, H., How was the notochord born? Evolution & Development 14:56-75. (2012)
(2)サンゴ礁の保全に関するゲノム科学的研究
海洋でサンゴ礁の占める面積は1%にも満たない。しかし、そこには全海洋生物種の約25%が棲息し、熱帯雨林と並んで地球上で最も生物多様性に富む場所とされている。一方、近年の海水温の上昇、海水の酸性化、オニヒトデの異常増殖などの環境悪化によって、世界中のサンゴ礁の存続が危機に瀕している。サンゴ礁の主体は造礁サンゴである。造礁サンゴの多くは受精後幼生の段階で褐虫藻の1群であるSymbiodiniumを体内に取り込んで共生関係をつくり、この共生関係に立脚して生きている。環境変化によるストレスなどで褐虫藻がサンゴからいなくなるといわゆる白化が起き、この状態が長引くとサンゴは死に至る。

(a) サンゴ礁に囲まれた沖縄島嶼は、サンゴ礁生物学を研究する上でまたとない研究の場を提供する。サンゴの生物学、特にサンゴと褐虫藻の共生関係の成立と崩壊の分子メカニズムに理解するためには、サンゴと共生褐虫藻の両方のゲノムを解読することが必須である。このような研究バックグランドのもとで、ユニット立ち上げ3年後の2011年に、我々は世界で初めてコユビミドリイシ(Acropora digitifera: 沖縄で最もよく見られる種で、1998年に起きたサンゴの大量白化死で最も被害の大きかったサンゴの1種)の約420 Mbpのゲノム解読に成功し、このサンゴが約24,000個の遺伝子を持つことを明らかにした[Shinzato et al., Nature 476: 320-323 (2011)]。さらに、造礁サンゴの祖先は化石記録から推定されるよりもかなり以前に分岐したと推定されること、コユビミドリイシを含むミドリイシサンゴ属はアミノ酸の1つシステインを自ら合成できない可能性が高いこと、サンゴは紫外線吸収物質MAAを作る遺伝子を1セット持っていること、共生と関連してか非常に複雑な自然免疫系を備えていること、などを明らかにした。
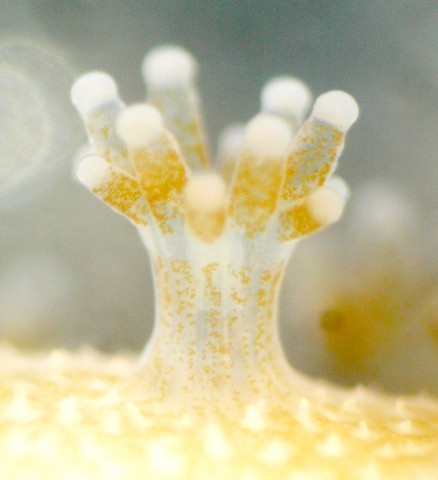 (b) 我々は続いてサンゴ共生褐虫藻の1種 Symbiodinium minutum(クレードBに属する)のゲノム解読に挑戦し、2013年に、これも世界に先駆けて、約1500 Mbpのゲノム解読に成功した(約42,000個の遺伝子)[Shoguchi et al., Curr. Biol. 23: 1399-1408 (2013)]。すなわち、OIST-MGUはサンゴと共生褐虫藻の両方のゲノムを世界で最初に解読したラボである。S. minutum のゲノムは予想されていたよりもはるかに複雑で、多くの遺伝子が一方向に並ぶという特徴に加えて、遺伝子は多くのイントロンで分断され、しかもイントロンの切り出しの分子メカニズムも通常の真核生物とかなり異なることが分かった。また、褐虫藻の染色体は常時凝縮しているという特徴を持つが、この凝縮に原核生物由来の遺伝子が多く使われていることも明らかになった。現在、サンゴに共生するクレードA, C, Dに属する褐虫藻のゲノム解読を進めており、また共生の成立と崩壊のメカニズムに関する研究も進めている。
(b) 我々は続いてサンゴ共生褐虫藻の1種 Symbiodinium minutum(クレードBに属する)のゲノム解読に挑戦し、2013年に、これも世界に先駆けて、約1500 Mbpのゲノム解読に成功した(約42,000個の遺伝子)[Shoguchi et al., Curr. Biol. 23: 1399-1408 (2013)]。すなわち、OIST-MGUはサンゴと共生褐虫藻の両方のゲノムを世界で最初に解読したラボである。S. minutum のゲノムは予想されていたよりもはるかに複雑で、多くの遺伝子が一方向に並ぶという特徴に加えて、遺伝子は多くのイントロンで分断され、しかもイントロンの切り出しの分子メカニズムも通常の真核生物とかなり異なることが分かった。また、褐虫藻の染色体は常時凝縮しているという特徴を持つが、この凝縮に原核生物由来の遺伝子が多く使われていることも明らかになった。現在、サンゴに共生するクレードA, C, Dに属する褐虫藻のゲノム解読を進めており、また共生の成立と崩壊のメカニズムに関する研究も進めている。
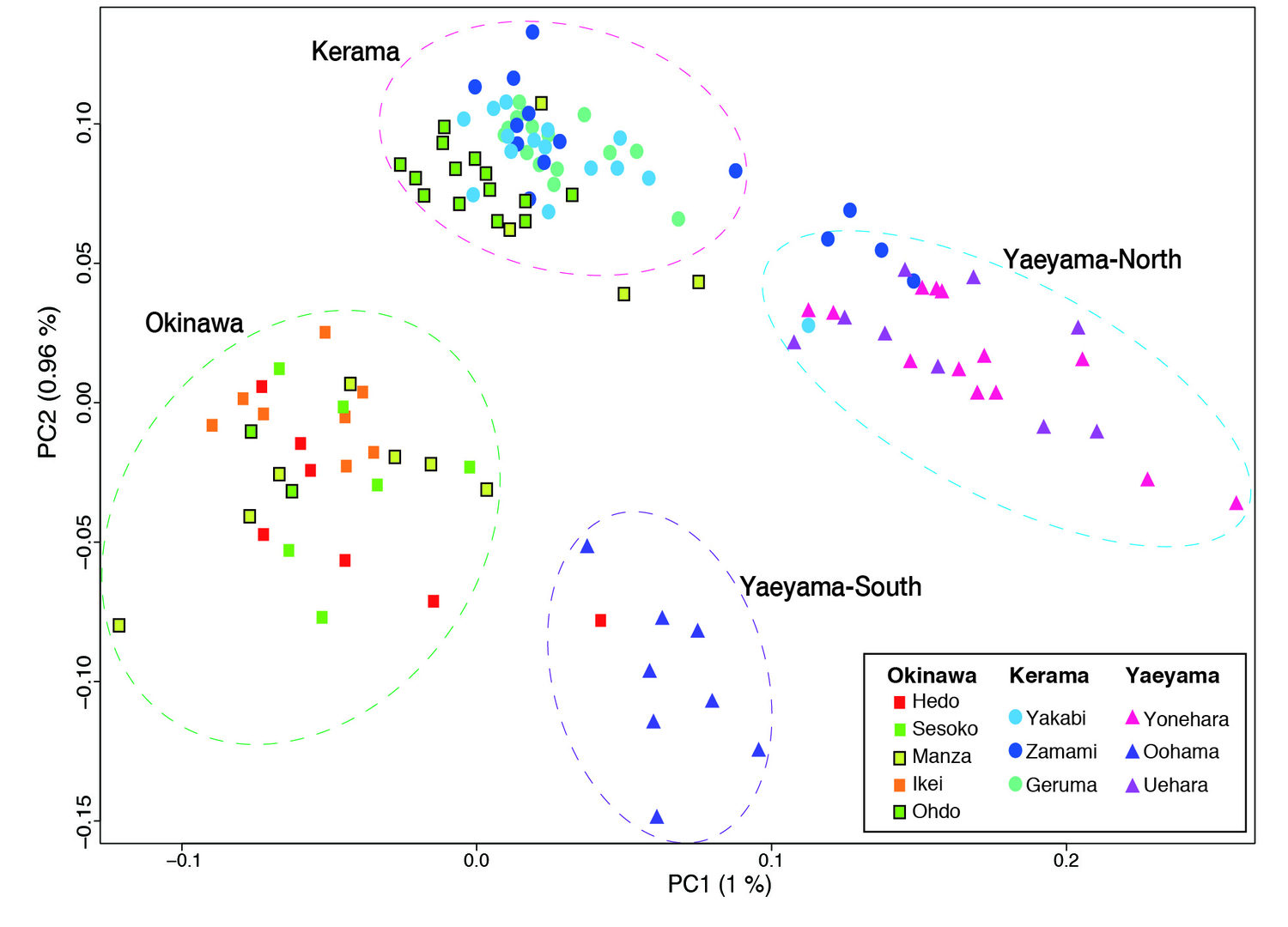 (c) 1998年のエルニーニョ現象によって世界中のサンゴ礁の多くが失われた。これは沖縄島嶼にも当てはまり、慶良間諸島を除く多くの島々でサンゴが白化し死滅した。しかしながら本島のサンゴ類はこの数年来徐々に回復傾向にある。これは慶良間諸島の自然産卵群が黒潮などの海流を利用して本島にたどり着き回復に寄与していると一般に考えられている。これらを検証するために、また沖縄島嶼におけるサンゴ礁の成り立ちを理解するために、八重山諸島、慶良間諸島、本島から約150群体におよぶコユビミドリを採集し、解読されたゲノム情報を駆使し1塩基多型レベルでの集団遺伝学的解析を行った[Shinzato et al., Sci. Reports, 5: 18211 (2015)]。その結果、図に示すように、沖縄島嶼のコユビミドリ集団は八重山北、八重山南、慶良間、沖縄本島の4つに分類され、慶良間諸島と沖縄本島の交流は本島南部(大渡など)のごく限られたものであることが判明した。すなわち、これまでの説とは異なり、回復しつつある本島のコユビミドリの多くは本島独自の群体に由来すると考える方が適切である。これは次の沖縄県のサンゴ礁保全再生事業に保全再生事業とも関連して重要な示唆を与える。
(c) 1998年のエルニーニョ現象によって世界中のサンゴ礁の多くが失われた。これは沖縄島嶼にも当てはまり、慶良間諸島を除く多くの島々でサンゴが白化し死滅した。しかしながら本島のサンゴ類はこの数年来徐々に回復傾向にある。これは慶良間諸島の自然産卵群が黒潮などの海流を利用して本島にたどり着き回復に寄与していると一般に考えられている。これらを検証するために、また沖縄島嶼におけるサンゴ礁の成り立ちを理解するために、八重山諸島、慶良間諸島、本島から約150群体におよぶコユビミドリを採集し、解読されたゲノム情報を駆使し1塩基多型レベルでの集団遺伝学的解析を行った[Shinzato et al., Sci. Reports, 5: 18211 (2015)]。その結果、図に示すように、沖縄島嶼のコユビミドリ集団は八重山北、八重山南、慶良間、沖縄本島の4つに分類され、慶良間諸島と沖縄本島の交流は本島南部(大渡など)のごく限られたものであることが判明した。すなわち、これまでの説とは異なり、回復しつつある本島のコユビミドリの多くは本島独自の群体に由来すると考える方が適切である。これは次の沖縄県のサンゴ礁保全再生事業に保全再生事業とも関連して重要な示唆を与える。
(d) 沖縄県は現在サンゴ礁を人為的に回復させようとする保全・再生事業を進めている。この際主として用いられるのがサンゴの枝を無性生殖的に増やし移植するという方法であるが、ここで問題になるのが、移植するサンゴの遺伝的多様性をどのように補完していくかである。そのためには、移植する大量のサンゴ群体の遺伝子的多様性をゲノムレベルで、かつ比較的簡単に調べる手法が必要である。我々は、コユビミドリイシのゲノム情報とこれにウスエダミドリイシのゲノム情報も加えて、14種のプライマーが1セットになったミドリイシ属サンゴの遺伝子的多様性を検出する分子生物学的手法を考案した[Shinzato et al., Front. Marine Sci. (2014)]。現在、この方法により、移植サンゴが遺伝的多様性を担保した形で行なわれるように、この事業に協力している。
 (e) オニヒトデ(Acanthaster planci)のゲノム解読:沖縄(OKI)では1970年頃からオニヒトデによるサンゴの食害が目立つようになり、主として人海戦術によってオニヒトデを駆除するという膨大な作業を行なってきた。同様の現象はオーストラリアのグレートバリアリーフ(GBR)でも起っており、この問題の生物学的解決に向けて豪日の共同研究がスタートした。そして2017年春にOKIおよびGBRそれぞれの1個体のゲノム(384 Mb)を解読した(Hall et al., Nature 544: 231-234 (2017) )。その結果、エペンデミン関連タンパク質を含むオニヒトデ個体間のコミュニケーションに関わると思われる分子をコードする遺伝子を数多く同定することに成功した。これらの情報を駆使してオニヒトデの同時大量捕獲に向けた研究の展開を計りたい。
(e) オニヒトデ(Acanthaster planci)のゲノム解読:沖縄(OKI)では1970年頃からオニヒトデによるサンゴの食害が目立つようになり、主として人海戦術によってオニヒトデを駆除するという膨大な作業を行なってきた。同様の現象はオーストラリアのグレートバリアリーフ(GBR)でも起っており、この問題の生物学的解決に向けて豪日の共同研究がスタートした。そして2017年春にOKIおよびGBRそれぞれの1個体のゲノム(384 Mb)を解読した(Hall et al., Nature 544: 231-234 (2017) )。その結果、エペンデミン関連タンパク質を含むオニヒトデ個体間のコミュニケーションに関わると思われる分子をコードする遺伝子を数多く同定することに成功した。これらの情報を駆使してオニヒトデの同時大量捕獲に向けた研究の展開を計りたい。
一方で、オニヒトデゲノムの解読は、その高品質の塩基配列アッセンブリーによって、棘皮動物を代表するゲノムとしてその解析が進んでいる。実際、このゲノム解析から、棘皮動物のHoxクラスターはHox6を失っているもののコリニアリテーを保っており、また(1)の(c)とも関連して、咽頭関連遺伝子クラスターも綺麗に保存されていることなどが分かってきている。現在、オニヒトデの発生学的特徴のゲノム科学的解析を進めている。加えて、5000km以上隔たった南半球GBRと北半球OKIの2個体のゲノム塩基配列が非常によく似かよっているという興味深い現象が明らかになった。これまでに得られている海産無脊椎動物のゲノム解析情報から判断すると、海底で棲息する動物が普通に集団を拡大していったと考えては説明が難しい現象である。オニヒトデの周期的大発生とも関連して、今後解析を進める必要がある。
参考文献
- Hall, M.R., Kocot, K.M., Baughman, K.W., Fernandez-Valverde, S.L., Gauthier, M.E.A., Hatleberg, W.L., Krishnan, A., McDougall, C., Motti, C.A., Shoguchi, E., Wang, T., Xiang, X., Zhao, M., Bose, U., Shinzato, C., Hisata, K., Fujie, M., Kanda, M., Cummins, S.F., Satoh, N., Degnan, S.M., Degnan. B.M. The crown-of-thorns starfish genome as a guide for biocontrol of this coral reef pest. Nature, 544: 231-234 (2017).
- Shinzato, C., Shoguchi, E., Kawashima, T., Hamada, M., Hisata, K., Tanaka, M., Fujie, M., Fujiwara, M., Koyanagi, R., Ikuta, T., Fujiyama, A., Miller, D. J., and Satoh, N. Using the Acropora digitifera genome to understand coral responses to environmental change. Nature, 476: 320-323 (2011).
- Shoguchi, E., Shinzato, C., Kawashima, T., Gyoja, F., Mungpakdee, S., Koyanagi, R., Takeuchi, T., Hisata, K., Tanaka, M., Fujiwara, M., Hamada, M., Azadeh, S., Fujie, M., Usami, T., Goto, H., Yamasaki, S., Arakaki, N., Suzuki, Y., Sugano, S., Toyoda, A., Kuroki, Y., Fujiyama, A., Medina, M., Coffroth, M. A., Bhattacharya, D., and Satoh, N. Draft assembly of the Symbiodinium minutum nuclear genome reveals dinoflagellate gene structure. Current Biology 23: 1399-1408 (2013).
- Shinzato, C., Mungpakdee, S., Arakaki, N., Satoh, N. Genome-wide SNP analysis explains coral diversity and recovery in the Ryukyu Archipelago. Scientific Reports 5, Article number: 18211 (2015) doi:10.1038/srep18211
- Shinzato, C., Yasuoka, Y., Mungpakdee, D., Arakaki, A., Fujie, M., Nakajima, Y., Satoh, N. Development of novel, cross-species microsatellite markers for Acropora corals using next-generation sequencing technology. Front. Mar. Sci., doi : 10.3389/fmars. 2014.00011 (2014).
(3)動物の特殊機能の獲得に関するゲノム科学的研究
(a)セルロースは最も大きなバイオマスであり、その効果的利用は地球環境と調和のとれた人間生活の維持にとっても重要である。セルロースの効果的利用にはセルロース生合成経路の分子メカニズムの理解が必要であるが、この合成は細胞膜上で起こる複雑な過程であるために、植物においてさえもそのメカニズムは解明されておらず、最近になってようやく細菌でそのメカニズムが解明されたに留まっている。ホヤ類は被嚢動物とも呼ばれ、セルロースを合成できる唯一の動物である。上述したカタユウレイボヤのゲノム解読の際に、この動物にはセルロース合成酵素遺伝子(CesA)が1コピー存在しか存在せず[Nakashima et al., Dev. Genes Evol. 214: 81-88 (2004)]、またトランスジェニック技術を駆使した研究で、この遺伝子が機能を失うとセルロースが合成されないことを確かめた[Sasakura et al., PNAS USA 102, 15134-15139 (2005)]。さらにオタマボヤでは、CesAが重複して2コピーになり、それぞれが結晶構造の異なるセルロース合成に関与することを発見した。現在、これらの研究成果をもとに、ホヤのセルロース生合成経路の分子メカニズムおよびin vivo合成系のさらなる解明を進めている。なお、この研究はOISTのPOC プログラムによって支援されている。
 (b) アコヤガイ(Pinctada fucata)は真珠をつくるという特殊機能を持つ。また真珠生産は日本の重要な産業の一つである。我々は国内の多くのアコヤガイ研究者と共同で2012年にアコヤガイゲノムを解読した[Takeuchi et al., DNA Res. 19: 117-130 (2012)]。さらにRNA情報などを加えたより良い遺伝子モデル ver2も2016年に発表した[Takeuchi et al., Zool. Letter 2: 3 (2016)]。現在、アコヤガイゲノム中に含まれるバイオミネラリゼション関連遺伝子の同定と、ゲノム情報を駆使したアコヤガイ系統の遺伝的バックグラウンドの探索を進めている。
(b) アコヤガイ(Pinctada fucata)は真珠をつくるという特殊機能を持つ。また真珠生産は日本の重要な産業の一つである。我々は国内の多くのアコヤガイ研究者と共同で2012年にアコヤガイゲノムを解読した[Takeuchi et al., DNA Res. 19: 117-130 (2012)]。さらにRNA情報などを加えたより良い遺伝子モデル ver2も2016年に発表した[Takeuchi et al., Zool. Letter 2: 3 (2016)]。現在、アコヤガイゲノム中に含まれるバイオミネラリゼション関連遺伝子の同定と、ゲノム情報を駆使したアコヤガイ系統の遺伝的バックグラウンドの探索を進めている。
 (c)我々は最近になって、より沖縄の水産業と関連したゲノム科学の展開の重要性を考え、1年ほど前からモズク(Cladosiphon okamuranus)およびウミブドウ(クビレズタ:Caulerpa lentillifera)のゲノム解読を、沖縄県水産海洋技術センターおよび恩納漁協の協力を得て進めている。その中でもモズクS株については約140 Mbのゲノム解読し、タンパク質をコードする約13,460遺伝子を同定して、2016年に公表した(Nishituji et al., DNA Res. 23: 561-570 (2016))。モズクはフコイダンを多く含む藻類として知られている。現在、フコイダン生合成経路に関わる酵素遺伝子の同定などを進めている。またウミブドウについては、この緑藻のゲノムサイズが約数十Mbと非常に小さいことなどが明らかになりつつある。
(c)我々は最近になって、より沖縄の水産業と関連したゲノム科学の展開の重要性を考え、1年ほど前からモズク(Cladosiphon okamuranus)およびウミブドウ(クビレズタ:Caulerpa lentillifera)のゲノム解読を、沖縄県水産海洋技術センターおよび恩納漁協の協力を得て進めている。その中でもモズクS株については約140 Mbのゲノム解読し、タンパク質をコードする約13,460遺伝子を同定して、2016年に公表した(Nishituji et al., DNA Res. 23: 561-570 (2016))。モズクはフコイダンを多く含む藻類として知られている。現在、フコイダン生合成経路に関わる酵素遺伝子の同定などを進めている。またウミブドウについては、この緑藻のゲノムサイズが約数十Mbと非常に小さいことなどが明らかになりつつある。
参考文献
- Nakashima, K., Yamada, L., Satou, Y., Azuma, J.-i. and Satoh, N.: The evolutionary origin of animal cellulose synthase. Dev. Genes Evol. 214, 81-88 (2004).
- Sasakura, Y., Nakashima, K., Awazu, S., Matsuoka, T., Nakayama, A., Azuma, J. and Satoh, N.: Transposon-mediated insertional mutagenesis revealed the functions of animal cellulose synthase in the ascidian Ciona intestinalis. Proc. Natl. Acad. Sci. USA 102, 15134-15139 (2005).
- Takeuchi, T., Kawashima, T., Koyanagi, R., Gyoja, F., Tanaka, M., Ikuta, T., Shoguchi, E., Fujiwara, M., Shinzato, C., Hisata, K., Fujie, M., Usami, T., Nagai, k., Maeyama, K., Okamoto, K., Aoki, H., Ishikawa, T., Masaoka, T., Fujiwara, A., Endo, K., Endo, H., Nagasawa, H., Kinoshita, S., Asakawa, S., Watabe, S., and Satoh, N., Draft Genome of the Pearl Oyster Pinctada fucata: A Platform for Understanding Bivalve Biology. DNA Research 19: 117-130. (2012)
- Takeuchi, T., Koyanagi, R., Gyoja, F., Kanda, M., Hisata, K., Fujie, M., Goto, H., Yamasaki, S., Nagai, K., Morino, Y., Miyamoto, H., Endo, K., Endo, H., Nagasawa, H., Kinoshita., S., Asakawa, S., Watabe, S., Satoh, N., Kawashima, T., Bivalve-specific gene expansion in the pearl oyster genome: Implications of adaptation to a sessile lifestylei. Zoological Letters, 2:3 (2016)
- Nishitsuji, K., Arimoto, A., Iwai, K., Sudo, Y., Hisata, K., Fujie, M., Arakaki, N., Kushiro, T., Konishi, T., Shinzato, C., Satoh, N., Shoguchi, E. A draft genome of the brown alga, Cladosiphon okamuranus, S-strain: a platform for future studies of 'mozuku' biology. DNA Research 23: 561-570 (2016).
(4)全動物門のゲノム解読に向けて
OISTはまだ新しい大学院大学であるが、ゲノム塩基配列の決定等についてはDNA Sequencing Section (SQC)の全面的支援を受けることができる。この機会を最大限に活用し、またこれから20年先の動物学への貢献を考え、国内外の多くの共同研究者の協力もあって、全動物門のゲノム解読に向けた研究も展開している。すでに述べたように、新口動物5群のゲノムは全て解読された。また、二胚葉動物についても全ての動物門のゲノム解読がなされており、残された門の多くはいわゆる旧口動物の螺旋動物群(Spiralia)あるいは冠論動物群(Lophotrochozoa)である。昨年に腕足動物シャミセンガイ(Ligula anatina)のゲノム解読に成功した。また箒虫動物と紐形動物のゲノム解読はほぼ完了しつつある。現在さらに、無腸動物、中生動物、毛顎動物、腹毛動物、内肛動物、外肛動物などのゲノム解読を目指している。
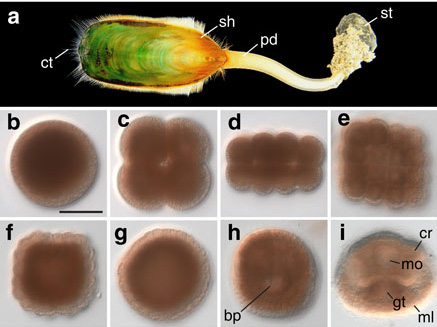 (a) 腕足動物シャミセンガイ(Ligula anatina)ゲノム。腕足動物は化石記録が豊富で、生きた化石とも呼ばれている。また現存する腕足動物と脊椎動物のみがリン酸カルシウムを用いたバイオミネラリゼションを行っており、進化的謎の一つとされている。我々は、東京大学と共同で、約425 MbのLigula anatinaゲノムを解読し、そこに含まれる約34,000遺伝子の解析を行った[Luo et al., Nature Communications 6: 8301 (2015)]。その結果、この動物の分子進化はこれまでゲノムが解読された動物の中で最も緩やかなものであり(生きた化石としての形態学的変化の少なさの要因)、また、この動物群のリン酸カルシウムを用いたバイオミネラリゼションは脊椎動物のものとは異なったメカニズムでなされていること、などが明らかになった。
(a) 腕足動物シャミセンガイ(Ligula anatina)ゲノム。腕足動物は化石記録が豊富で、生きた化石とも呼ばれている。また現存する腕足動物と脊椎動物のみがリン酸カルシウムを用いたバイオミネラリゼションを行っており、進化的謎の一つとされている。我々は、東京大学と共同で、約425 MbのLigula anatinaゲノムを解読し、そこに含まれる約34,000遺伝子の解析を行った[Luo et al., Nature Communications 6: 8301 (2015)]。その結果、この動物の分子進化はこれまでゲノムが解読された動物の中で最も緩やかなものであり(生きた化石としての形態学的変化の少なさの要因)、また、この動物群のリン酸カルシウムを用いたバイオミネラリゼションは脊椎動物のものとは異なったメカニズムでなされていること、などが明らかになった。
またOIST-MGUの一つの特徴は、ラボの研究員の幾人かは、上記のユニットのメイン研究テーマとは関係なく、独自のテーマで海産無脊椎動物の発生と進化などに関する研究を進めていることである。この研究には、刺胞動物の発生における形態形成のメカニズム、ハゼ科魚類の生態地理学的研究などが含まれる。
参考文献
- Luo, Y.J., Takeuchi, T., Koyanagi, R., Yamada, L., Kanda, M., Khalturina, M., Fujie, M., Yamasaki, S., Endo, K., Satoh, N.,The Lingula genome provides insights into brachiopod evolution and the origin of phosphate biomineralization. Nature Communications, 18;6:8301. doi: 10.1038/ncomms9301. (2015)
ニュースフィード
以下は本年度のユニットの研究成果で、OISTニュースフィードで紹介されたものです。
詳細はニュースフィードのそれぞれの記事を参照して下さい。